
信息整理:图书馆
2022年诺贝尔生理学或医学奖被授予瑞典科学家斯万特·佩博(Svante Pääbo),以表彰他在已灭绝古人类基因组和人类进化研究方面做出的贡献。以下对其相关主题学术论文和相关施引文献进行分析。
一、获奖者的发文分析
斯万特·佩博的SCIE/SSCI论文共计313篇(Article/Review),其中,相关主题论文139篇,发文的年度分布如图1所示。
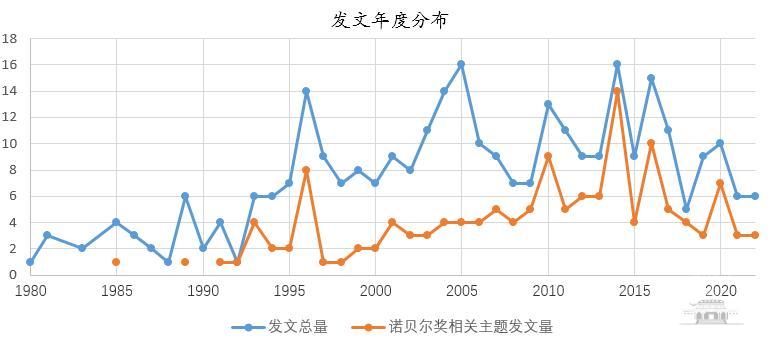
图1 SCIE/SSCI论文的年度分布
这139篇论文涉及分子生物学和遗传学、生物和生物化学等学科领域,论文分布于37种期刊,其中,49篇发表在Cell、Nature和Science期刊上,占139的36.25%。
获奖者的相关主题学术论文详见武汉大学图书馆医学学科服务平台:https://libguides.lib.whu.edu.cn/c.php?g=665808&p=4682170
获奖者的313篇SCIE/SSCI论文中,由于数据库数据不全,有4篇早期论文不在分析之列 ;表1是其余309篇论文所涉及的13个ESI学科领域及各组论文的学科影响力表现 ;不难看出,获奖者在分子生物学与遗传学领域贡献突出,论文数占全部论文的64.2%,篇均被引高达228.77次。
表1 SCIE/SSCI论文归属学科及影响力表现
ESI学科
论文数
被引频次
篇均被引
学科规范化的引文影响力
排名前 1% 的论文
平均百分位
Molecular Biology & Genetics
201
45983
228.77
4.23
23
80.70
Biology & Biochemistry
56
7908
141.21
3.14
5
73.87
Social Sciences, general
10
1225
122.5
5.22
2
77.80
Neuroscience & Behavior
12
1224
102
2.94
1
83.49
Multidisciplinary
5
1053
210.6
7.40
0
95.87
Environment/Ecology
6
615
102.5
2.28
0
81.07
Plant & Animal Science
2
515
257.5
9.35
1
99.18
Clinical Medicine
10
306
30.6
0.89
0
55.37
Immunology
2
146
73
1.51
0
63.75
Computer Science
1
134
134
4.63
0
96.76
Geosciences
2
94
47
3.01
0
94.46
Microbiology
1
26
26
7.29
0
98.68
Pharmacology & Toxicology
1
0
0
0
0
0.0031
139篇诺贝尔奖相关主题论文涉及9个ESI学科,其中分子生物学与遗传学领域论文96篇,占139的69%,篇均被引高达273.57次,各组论文表现见表2。
表2 诺贝尔奖相关主题论文归属学科及影响力表现
ESI学科
论文数
被引频次
篇均被引
学科规范化的引文影响力
排名前 1% 的论文
平均百分位
Molecular Biology & Genetics
96
26263
273.57
5.50
15
86.22
Biology & Biochemistry
20
4412
220.6
4.91
4
84.20
Social Sciences, general
8
1176
147
6.12
2
79.42
Multidisciplinary
5
1053
210.6
7.40
0
95.87
Neuroscience & Behavior
5
498
99.6
2.45
0
84.84
Plant & Animal Science
1
171
171
6.49
0
98.70
Geosciences
2
94
47
3.01
0
94.46
Clinical Medicine
1
26
26
0.74
0
65.02
Environment/Ecology
1
2
2
0.51
0
42.38
通过对139篇相关主题文献的全部关键词进行词频统计、得到高频关键词词云,主要高频词有:Sequence(44)、Ancient DNA(41)、Evolution(32)、Genome Sequence(29)、Neanderthal(26)、Human-Evolution(21)等,如图2所示。
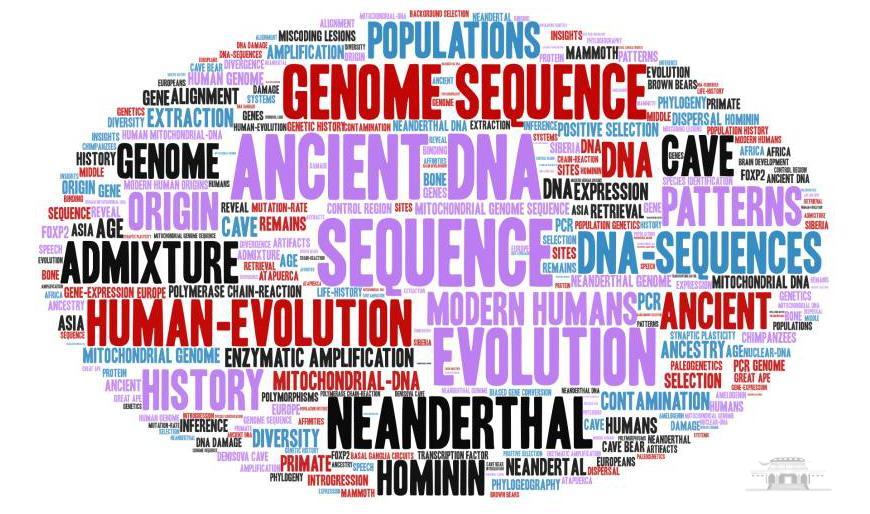
图2 相关主题文献的高频关键词词云
二、相关施引文献分析
截至2022年10月8日,斯万特·佩博的139篇相关文献被全球16439篇论文引用,总被引33717次,篇均被引242.57次;从全球来看,施引文献逐年持续增长,1985年仅5篇、2020年高达1638篇。全部施引文献分布在180个国家/地区 ;排名第一的是美国、发文量达6825篇 ;其次是英国、2676篇,第三是德国、2523篇 ;中国排在第六、发文量为1258篇。
施引文献涉及的研究领域比较广泛;我们结合本次诺奖的获奖原因,构造相应主题检索策略,从施引文献中筛选出与诺奖主题相关的论文共计4610篇,下面从不同角度分析这些相关施引文献。
1.发文年度分布
相关施引文献量呈逐年上升趋势,如图3所示,1989年3篇,2021年高达403篇。

图3 相关施引文献年度分布
2.发文期刊分布
4610篇相关施引文献分布在834种期刊,发文较多的 20 种期刊刊登相关论文1935篇、占全部相关主题论文的42%。TOP20期刊见图4。
3.发文机构分布
4610篇论文涉及全球156个国家/地区,其中,中国作者参与的相关施引文献共计310篇,论文数≥5篇的中国机构如图5所示。
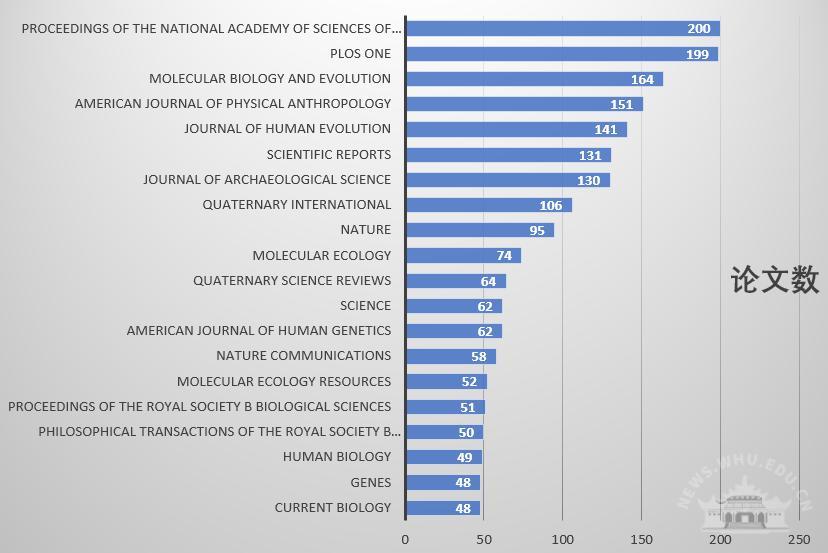
图4 相关施引文献期刊分布(TOP 20)
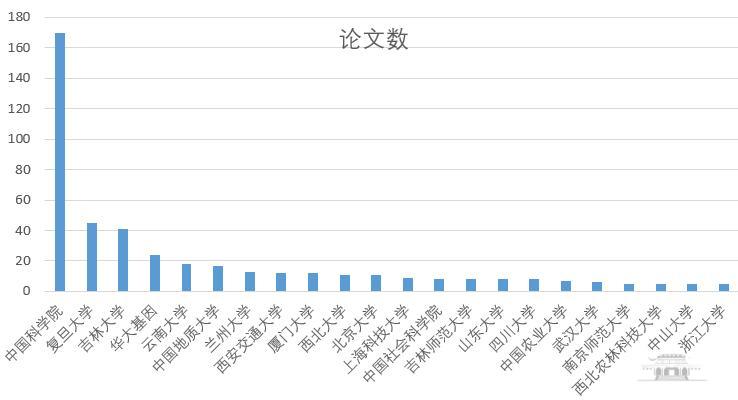
图5 相关施引文献中论文数≥5篇的中国机构
4.引文主题领域分布
基于 Citation Topics的宏观领域,4610篇相关主题论文涉及9个领域,主要集中在Clinical & Life Sciences(2877篇,占62.40%)、Earth Sciences(1032篇,占22.39%)和Agriculture, Environment & Ecology(560篇,占12.15%)三个领域。
中国作者参与的310篇论文涉及5个领域:主要集中在Clinical & Life Sciences(227篇,占89.35%)、Earth Sciences(58篇,占18.70%)和Agriculture, Environment & Ecology(16篇,占5.16%)三个领域。
基于 Citation Topics 的中观领域,4610篇相关主题研究论文涉及122个中观领域,主要集中在Genome Studies(2433篇,占52.78%)、Archaeology(1008篇,占21.87%)和Phylogenetics & Genomics(310篇,占6.72%)等领域。
中国作者参与的310篇论文涉及27个中观领域,主要集中在Genome Studies(203篇,占65.48%)、Archaeology(56篇,占18.06%)和Phylogenetics & Genomics(8篇,占2.58%)等领域。
基于 Citation Topics 的微观领域,4610篇相关研究论文涉及257个微观领域,主要集中在Population Genetics(2414篇,占52.36%)、Holocene(485篇,占10.52%)和Mammalia(332篇,占7.20%)等领域。
中国作者参与的310篇涉及37个微观领域,主要集中在 Population Genetics(202篇,占65.16%)、Holocene(28篇,占9.03%)和Mammalia(24篇,占7.74%)等领域。
上述数据表明,中国参与的相关论文与全球的相关论文从引文主题角度比较来看基本一致。
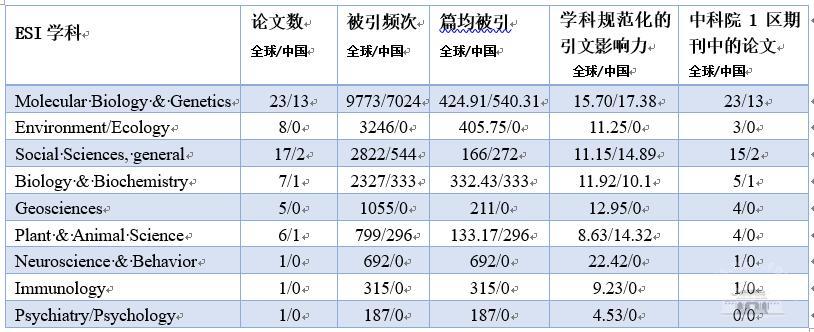
5.相关施引文献的高被引论文学科分布
相关施引文献中,近十年有高被引论文69篇,其中中国作者参与的有17篇,表3是高被引论文中全球和中国作者参与的论文涉及的ESI学科及各组论文影响力表现比较。
表3 高被引论文归属学科及影响力表现
6.相关施引文献高频关键词
通过对4610篇相关主题施引文献关键词进行词频统计、得到高频关键词词云,主要高频词有:Ancient DNA(2032)、Sequence(977)、Neanderthal(864)、Evolution(733)、DNA-Sequences(621)、Human-Evolution(519)等,如图6所示。对比获奖者相关文献词频分析,可发现二者的前6位高频词中有5个词是相同的;唯一不同的词是施引文献中的DNA-Sequences和获奖者文献中的Genome Sequence。

图6 相关主题施引文献高频关键词词云
三、相关主题高影响力论文
斯万特·佩博的139篇相关主题论文中,影响力高的文献较多,最高的被引次数达到2265次,发表于2010年;近十年高被引论文12篇,3篇Nature发文的第一作者是中国科学院古脊椎动物与古人类研究所的付巧妹,见[3] [4] [5] ;有9篇论文有中国作者参与研究;基于ESI高被引仅显示近十年的论文,以下列举20篇高影响力论文供参考。
[1] Zeberg H, Paabo S.The major genetic risk factor for severe COVID-19 is inherited from Neanderthals[J]. Nature, 2020, 587(7835): 610.
[2] Mallick S, Li H, Lipson M, et al.The Simons Genome Diversity Project: 300 genomes from 142 diverse populations[J]. Nature, 2016, 538(7624): 201.
[3] Fu Q M, Posth C, Hajdinjak M, et al.The genetic history of Ice Age Europe[J]. Nature, 2016, 534(7606): 200.
[4] Fu Q, Hajdinjak M, Moldovan O T, et al.An early modern human from Romania with a recent Neanderthal ancestor[J]. Nature, 2015, 524(7564): 216.
[5] Fu Q M, Li H, Moorjani P, et al.Genome sequence of a 45,000-year-old modern human from western Siberia[J]. Nature, 2014, 514(7523): 445.
[6] Lazaridis I, Patterson N, Mittnik A, et al.Ancient human genomes suggest three ancestral populations for present-day Europeans[J]. Nature, 2014, 513(7518): 409.
[7] Sankararaman S, Mallick S, Dannemann M, et al.The genomic landscape of Neanderthal ancestry in present-day humans[J]. Nature, 2014, 507(7492): 354.
[8] Meyer M, Fu Q M, Aximu-Petri A, et al.A mitochondrial genome sequence of a hominin from Sima de los Huesos[J]. Nature, 2014, 505(7483): 403.
[9] Prufer K, Racimo F, Patterson N, et al.The complete genome sequence of a Neanderthal from the Altai Mountains[J]. Nature, 2014, 505(7481): 43.
[10] Dabney J, Knapp M, Glocke I, et al.Complete mitochondrial genome sequence of a Middle Pleistocene cave bear reconstructedfrom ultrashort DNA fragments[J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(39): 15758-15763.
[11] Fu Q M, Mittnik A, Johnson P, et al.A RevisedTimescale for Human Evolution Based on Ancient Mitochondrial Genomes[J]. Current Biology, 2013, 23(7): 553-559.
[12] Meyer M, Kircher M, Gansauge M T, et al.A High-Coverage Genome Sequence from an Archaic Denisovan Individual[J]. Science, 2012, 338(6104): 222-226.
[13] Reich D, Green R E, Kircher M, et al.Genetic history of anarchaic hominin group from Denisova Cave in Siberia[J]. Nature, 2010, 468(7327): 1053-1060.
[14] Green R E, Krause J, Briggs A W, et al.A Draft Sequence of the Neandertal Genome[J]. Science, 2010, 328(5979): 710-722.
[15] Mikkelsen T S, Hillier L W, Eichler E E, et al.Initial sequence of the chimpanzee genome and comparison with the human genome[J]. Nature, 2005, 437(7055): 69-87.
[16] Paabo S, Poinar H, Serre D, et al.Genetic analyses from ancient DNA[J]. Annual Review of Genetics, 2004, 38: 645-679.
[17] Enard W, Przeworski M, Fisher S E, et al.Molecular evolution of FOXP2, a gene involved in speech and language[J]. Nature, 2002, 418(6900): 869-872.
[18] Ingman M, Kaessmann H, Paabo S, et al.Mitochondrial genome variation and the origin of modern humans[J]. Nature, 2000, 408(6813): 708-713.
[19] Krings M, Stone A, Schmitz R W, et al.Neandertal DNA sequences and the origin of modern humans[J]. Cell, 1997, 90(1): 19-30.
[20] Paabo S.Ancient DNA - extraction, characterization, molecular-cloning, and enzymatic amplification[J]. Proceedings of the National Academy of Sciences of the United States of America, 1989, 86(6): 1939-1943.
四、高影响力施引文献
在2020年以来的相关施引文献中,ESI高被引论文13篇,论文信息如下:
[1] Prevost M, Groman-Yaroslavski I, Gershtein K, et al.Early evidence for symbolic behavior in the Levantine Middle Paleolithic: A 120 ka old engravedaurochs bone shaft from the open-air site of Nesher Ramla, Israel[J]. Quaternary International, 2022, 624: 80-93.
[2] Doan K, Niedzialkowska M, Stefaniak K, et al.Phylogenetics and phylogeography of red deer mtDNA lineages during the last 50 000 years in Eurasia[J]. Zoological Journal of the Linnean Society, 2022, 194(2): 431-456.
[3] Slimak L, Zanolli C, Higham T, et al.Modernhumanincursion into Neanderthal territories 54,000 years ago at Mandrin, France[J]. Science Advances, 2022, 8(6).
[4] Huffman J E, Butler-Laporte G, Khan A, et al.Multi-ancestry fine mapping implicates OAS1 splicing in risk of severe COVID-19[J]. Nature Genetics, 2022, 54(2): 125.
[5] Hershkovitz I, May H, Sarig R, et al.A Middle Pleistocene Homo from Nesher Ramla, Israel[J]. Science, 2021, 372(6549): 1424.
[6] Wibowo M C, Yang Z, Borry M, et al.Reconstruction of ancient microbial genomes from the human gut[J]. Nature, 2021, 594(7862): 234.
[7] Zhou S R, Butler-Laporte G, Nakanishi T, et al.A Neanderthal OAS1 isoform protects individuals of European ancestry against COVID-19 susceptibility and severity[J]. Nature Medicine, 2021, 27(4).
[8] Wang C C, Yeh H Y, Popov A N, et al.Genomicinsights into the formation of human populations in East Asia[J]. Nature, 2021, 591(7850): 413.
[9] van der Valk T, Pecnerova P, Diez-Del-Molino D, et al.Million-year-old DNA sheds light on the genomic history of mammoths[J]. Nature, 2021, 591(7849): 265.
[10] Bergstrom A, Stringer C, Hajdinjak M, et al.Origins of modern human ancestry[J]. Nature, 2021, 590(7845): 229-237.
[11] Zeberg H, Paabo S.The major genetic risk factor for severe COVID-19 is inherited from Neanderthals[J]. Nature, 2020, 587(7835): 610.
[12] Welker F, Ramos-Madrigal J, Gutenbrunner P, et al.The dental proteome of Homo antecessor[J]. Nature, 2020, 580(7802): 235.
[13] Bergstrom A, Mccarthy S A, Hui R Y, et al.Insights into human genetic variation and population history from 929 diverse genomes[J]. Science, 2020, 367(6484): 1339.
以上内容由大学时代综合整理自互联网,实际情况请以官方资料为准。